- 塩基配列の取得
[GenBank]
- ORFの検索
[ORF Finder]
- SD配列の検索
[GeneMark]
- プロモーター領域の予測
[Neural Network Promoter]
- 転写終結部位予測
[FindTerm]
- 塩基配列の諸性質
[MBCF OligoCalculator]
- アミノ酸配列の取得
- タンパク質の諸性質
[ProtParam]
- 類似性検索
[BLAST]
- モチーフ配列検索
[MOTIF]
- 系統樹の作成
[UniProt, ClustalW]
- ヒトゲノムとの相同性解析とマッピング
[BLAST]
- 文献検索
[PubMed]
- 膜貫通領域の予測
[SOSUI]
- 二次構造予測
[SSP]
- 三次構造予測
[SWISS-MODEL]
- 構造を見る
[PDB]
実習のデータ
実習の検索結果のデータです。
これは、はじめから使わないで、はじめは自分でデータをとるようにしましょう。
(山登よりのお願い。インターネットの通信混雑と検索側計算機の負荷軽減にも ご配慮下さい。その意味で、田中君にお願いして、ここに検索結果のデータを載せて 頂きました。少なくともこの実習でインターネットをご利用になるときは、自分の 成長に資することを目的にしているということを、しっかり念頭に置いてご利用下さい。)
バイオインフォマティクスの基礎実習
- V-ATPase遺伝子群塩基配列の取得
① DBGET(データベース統合検索システム)にアクセスし、GenBank をクリックする。
② GenBankの入力枠内に "Enterococcus hirae ntp operon" とキーワードを入力し、Go ボタンをクリックする。
③ 検索結果の gb:ENENTP をクリックし、開かれたページを "お気に入り" に登録しておく(以下、このページをGenBank resultページと呼ぶ)。
 イオン輸送性 ATPase について
イオン輸送性 ATPase について
- ORF (Open Reading Frame) の検索
① GenBank resultページにある、FASTA をクリックし、FASTA形式のDNAシークエンスを表示させる。
② ORF Finderの入力枠内に、このDNAシークエンスをコピー&ペーストし、OrfFind ボタンをクリックする。
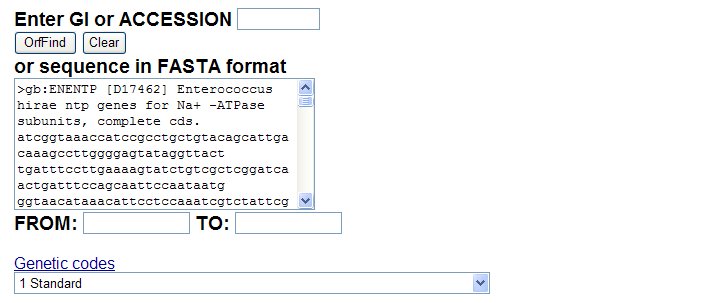
③ GenBank resultページの CDS 行を参考に、サブユニットF・I・K・E・C・G・A・B・D・H・Jに対応する ORF をクリックし、表示された塩基配列・アミノ酸配列と比較し、良ければ Accept ボタンを押す。
【例、E サブユニットの場合】
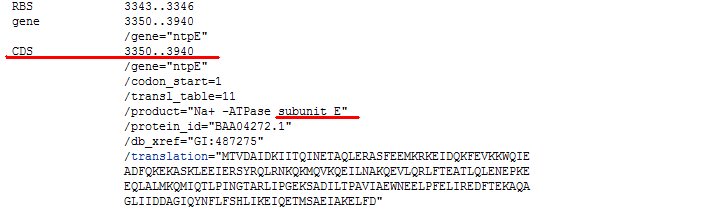
↓
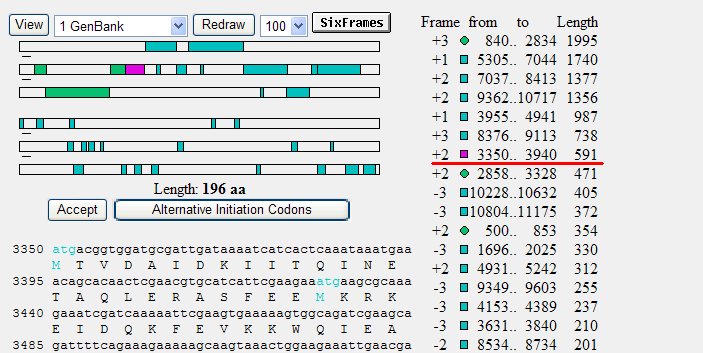
④ サブユニットの中には終止コドンのみが一致するものがある。その際は、Alternative Initiation Codons をクリックし、表示された結果が正しければ、Accept ボタンを押す。
【注】このページは後で使用するため、閉じずにそのままにしておく。
【TOPへ戻る】
- SD (Shine-Dalgarno) 配列の検索
GeneMarkの Sequence の枠内にDNAシークエンスをコピー&ペーストする。Species は Enterococcus_faecalis を選択し、Start GeneMarkボタンをクリックする。
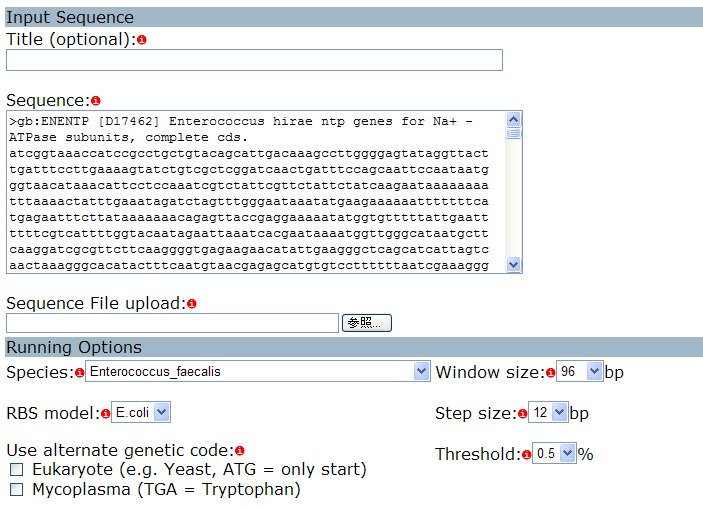
【TOPへ戻る】
- プロモーター領域の予測
Neural Network Promoterの Type of organismはprokaryote、Include reverse strand?はnoを、選択スコアはデフォルトのまま、Cut and paste your sequence(s) here: の枠内にFASTA形式のDNAシークエンスをコピー&ペーストし、Submit ボタンをクリックする。
【TOPへ戻る】
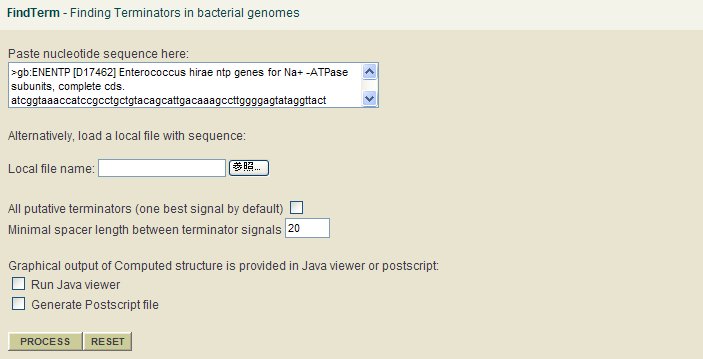
- 転写終結部位の推定
FindTermの Paste nucleotide sequence here: の枠内にDNAシークエンスをコピー&ペーストし、PROCESS ボタンをクリックする。
【TOPへ戻る】
- DNA塩基配列の諸性質
Oligo Calculatorの入力枠内に、A, B, I の各サブユニットをコードするORFのDNAシークエンスをコピー&ペーストし、Calculate ボタンをクリックする。
【TOPへ戻る】
- A サブユニットと B サブユニットのアミノ酸配列の取得
ORF finderのページの、Viewの横の選択Boxで3 Fasta protein を選択し、FASTA形式のアミノ酸配列を表示させる。
subunit A, subunit B についてアミノ酸配列を取得する。
【TOPへ戻る】
- タンパク質の諸性質
ProtParamの入力枠内に、A サブユニットのアミノ酸配列(FASTA形式の第1行目は入れない)をコピー&ペーストし、Compute parameters ボタンをクリックする。
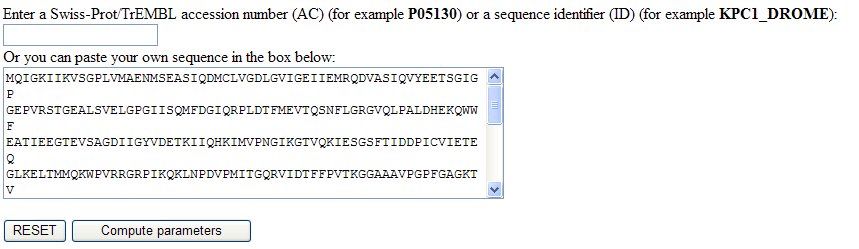
同様に B サブユニットについても調べる。
【TOPへ戻る】
- タンパク質のアミノ酸配列の類似性検索
BLASTの入力枠内に、A サブユニットのアミノ酸配列をコピー&ペーストする。
プログラムは BLASTP (prot query vs prot db) 、データベースは KEGG GENES を選択し、Compute ボタンをクリックする。
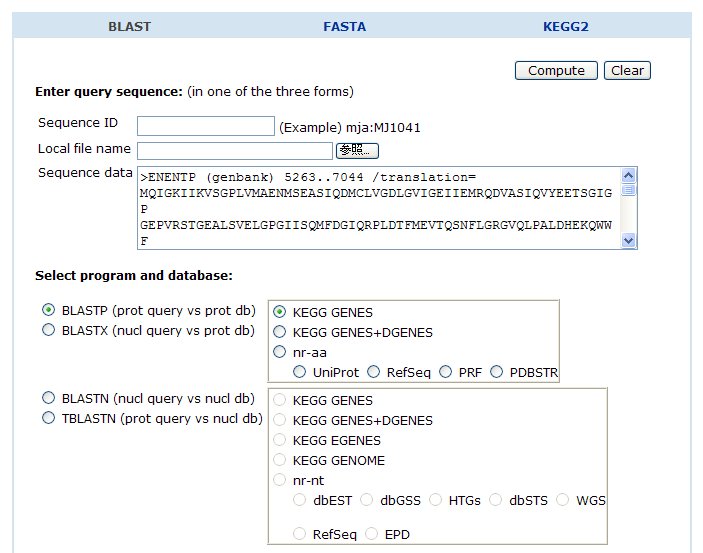
同様に B サブユニットについても調べる。
 BLAST について
BLAST について
【TOPへ戻る】
- モチーフ配列の検索
MOTIFの入力枠内に、A サブユニットのアミノ酸配列をコピー&ペーストする。
For protein をチェックし、データベースは PROSITE Pattern を選択し、Skip entries with SKIP-FLAG のチェックははずして、Search ボタンをクリックする。
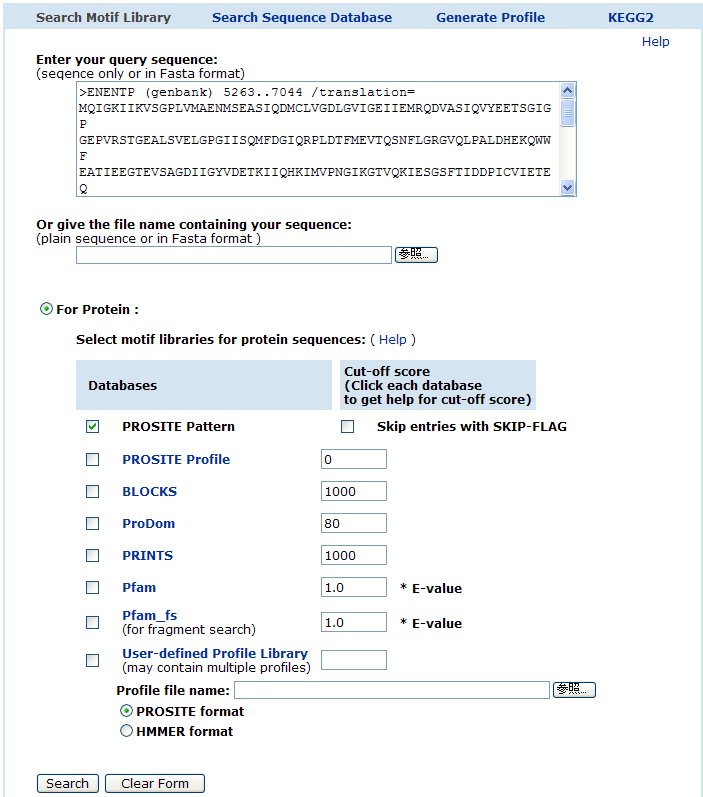
同様に B サブユニットについても調べる。
【TOPへ戻る】
- 系統樹の作成
① DBGET(データベース統合検索システム)にアクセスし、UniProt をクリックする。
② キーワードとして、V-ATPase "subunit A" を入力し、Go ボタンをクリックする。
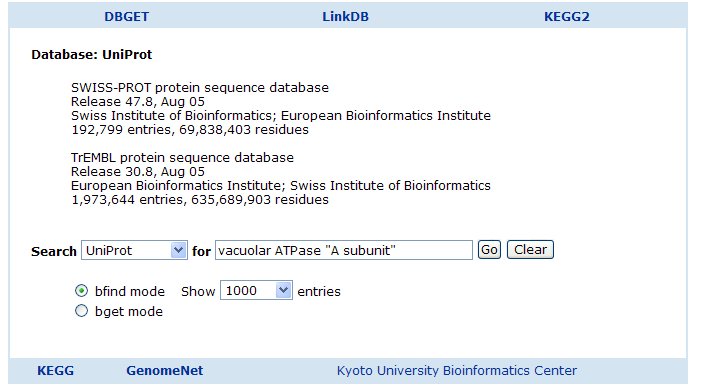
③ 作業④を、[sp:VATA_HUMAN]、[sp:VATA_PIG]、[sp:VATA_BOVINE]、[sp:VATA_MOUSE]、[sp:VATA1_DROME]、[sp:VATA_SCHPO]、[sp:VATA_YEAST]、[sp:VATA1_ACEAT] の8種のタンパク質について行う。それぞれのタンパク質が何の生物種由来かは、各自調べること。
④ sp:VATA_HUMAN を例とする。
sp:VATA_HUMAN をクリックする。
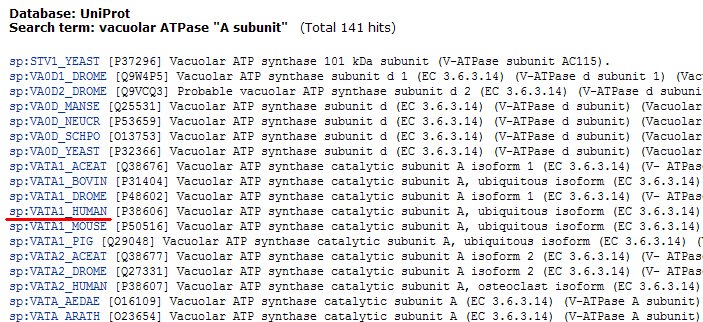
下の方にある SQ をクリックする。
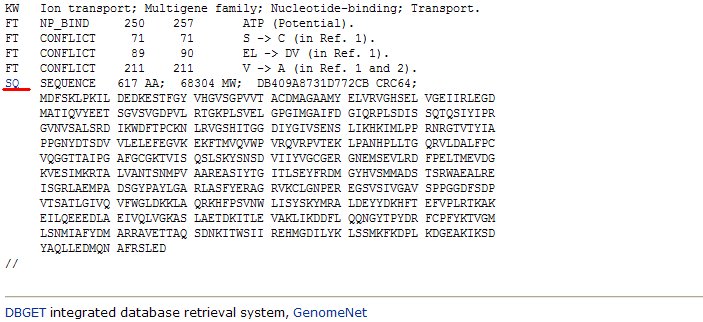
表示されたアミノ酸配列を、メモ帳にコピー&ペーストし、先頭行を >HUMAN に書き換える。
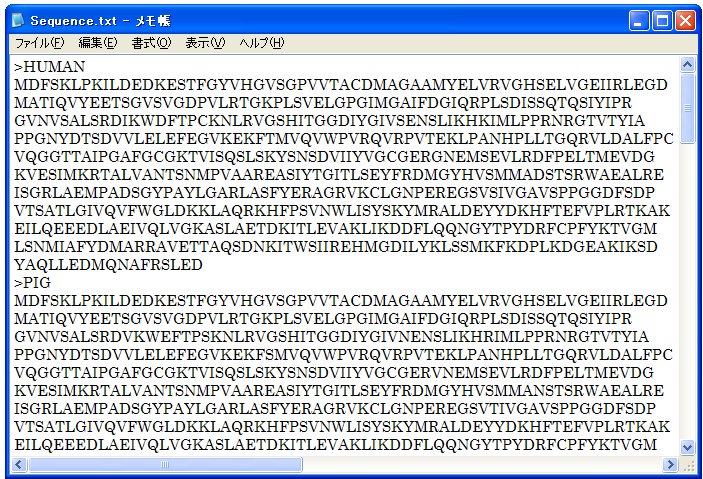
⑤ 上の図のように8種のアミノ酸配列を、1つのメモ帳にまとめて保存する。
⑥ 最後に、GenBank result のページから、腸内連鎖球菌V-ATPase のAサブユニットのアミノ酸配列をコピーし、メモ帳にペーストして、先頭行を >ENTERO に書き換える。
⑦ Clustal Wの入力枠内に、作成した9種のタンパク質のアミノ酸配列情報をコピー&ペーストし、Execute Multiple Alignment のボタンをクリックする。。
⑧ 一番下のSelect tree menu のタブから、N-J Tree を選択し、Exec ボタンをクリックすると、系統樹が表示される。
【TOPへ戻る】
- ヒトゲノムとの相同性解析とマッピング
① NCBI Human Genome Resourceにアクセスし、BLAST the Genome クリックする。
データベースをRefSeq protein、プログラムをBLASTP: Compare protein sequences とし、入力枠内にAサブユニットのアミノ酸配列をコピー&ペーストし、Begin Search のボタンをクリックする。
② 次のページでFormat のボタンをクリックし、少し待つと結果が表示される。
③ 検索の結果、相同性の高いタンパク質がいくつか見つかるが、その中で最も相同性の高い human の V-ATPase に注目する。
④ human の V-ATPase の右の G をクリックし、表示されたページの ATP6V1A をクリックして、このタンパク質についての情報を得る。
特に、染色体番号、場所、機能について注目する。
⑤ ページ右の Map Viewer をクリックし、染色体上の位置を確認する。
【TOPへ戻る】
- 文献検索
V-ATPase に異常があるとどのような病気になるのかを論文で調べる。
Entrez PubMedで、キーワードとして、「vacuolar ATPase human disease mutation」 を入力し Go ボタンをクリックすると、検索された論文の一覧が表示される。

【TOPへ戻る】
- 水溶性タンパク質か膜タンパク質かの予測
① SOSUIの1. SOSUI system の SOSUI をクリックする。入力枠内に、A サブユニットのアミノ酸配列(FASTA形式の第1行目は入れない)をコピー&ペーストし、Exec ボタンをクリックする。
② 同様に、B サブユニット、I サブニユットについても予測する。
【TOPへ戻る】
- 二次構造の予測
SSPのChou-Fasman, GOR, Neural Networkすべてを選択し、Paste the amino acid sequence in the box:枠内に、A サブユニットのアミノ酸配列(FASTA形式の第1行目は入れない)をコピー&ペーストし、start prediction ボタンをクリックする。
同様に、B サブユニット、I サブニユットについても予測する。
【TOPへ戻る】
- 三次構造の予測
【注】三次構造の予測は計算量が多く、結果を出すまでに時間がかかるため、結果は E-mail で返信される。よって、実習では計算結果をこちらで配布する。ただし、自宅でインターネットに接続できる人は、ぜひ作業を行ってほしい。
SWISS-MODELの First Approach mode をクリックする。Email アドレス、名前、query のタイトル(例: V-ATPase subunit A)、A サブユニットのアミノ酸配列を入力枠内にコピー&ペーストする。ページの下部にある Results options は Normal mode にし、Send Request ボタンをクリックする。
同様に、B サブユニット、K サブニユットについても予測する。
【TOPへ戻る】
- ホモロジーの高い構造を見る
① SWISS-MODELのMENU の Interactive tools の項目にある Search をクリックする。
② FASTA 形式の A サブユニットのアミノ酸配列を入力枠内にコピー&ペーストし、Submit Request ボタンをクリックする。
③ 表示された結果から 1bmfF の Detail をクリックし、特徴を調べる。
また、Parent PDB 列の 1bmf をクリックし、Protein Data Bank (PDB) にアクセスする。

④ Download / Display File をクリックし、Download the Structure File: の ZIPped の X をクリックし、コンピュータの適当なところにファイルを保存する。
また、View Structure をクリックし、Interactive 3D Display: の QuickPDB ボタンをクリックしたり、Still Images: のリンクをクリックして、1bmf の立体構造を見る。
⑤ 同様に、B サブユニット、K サブニユットについても調べる。
検索結果から、 B サブユニットでは、1bmfF、K サブユニットでは 2bl2 について調べる。
【TOPへ戻る】