研究室の概要
当研究室は、東京理科大学理学部化学系研究室の中で唯一の理論系研究室として、「化学」を主軸に「生物」「物理」「計算」「情報」をスーパーコンピュータと AI で融合させる次世代理論化学研究を目指し、主に生物化学、創薬化学、環境化学に関する問題に取り組んでいます。
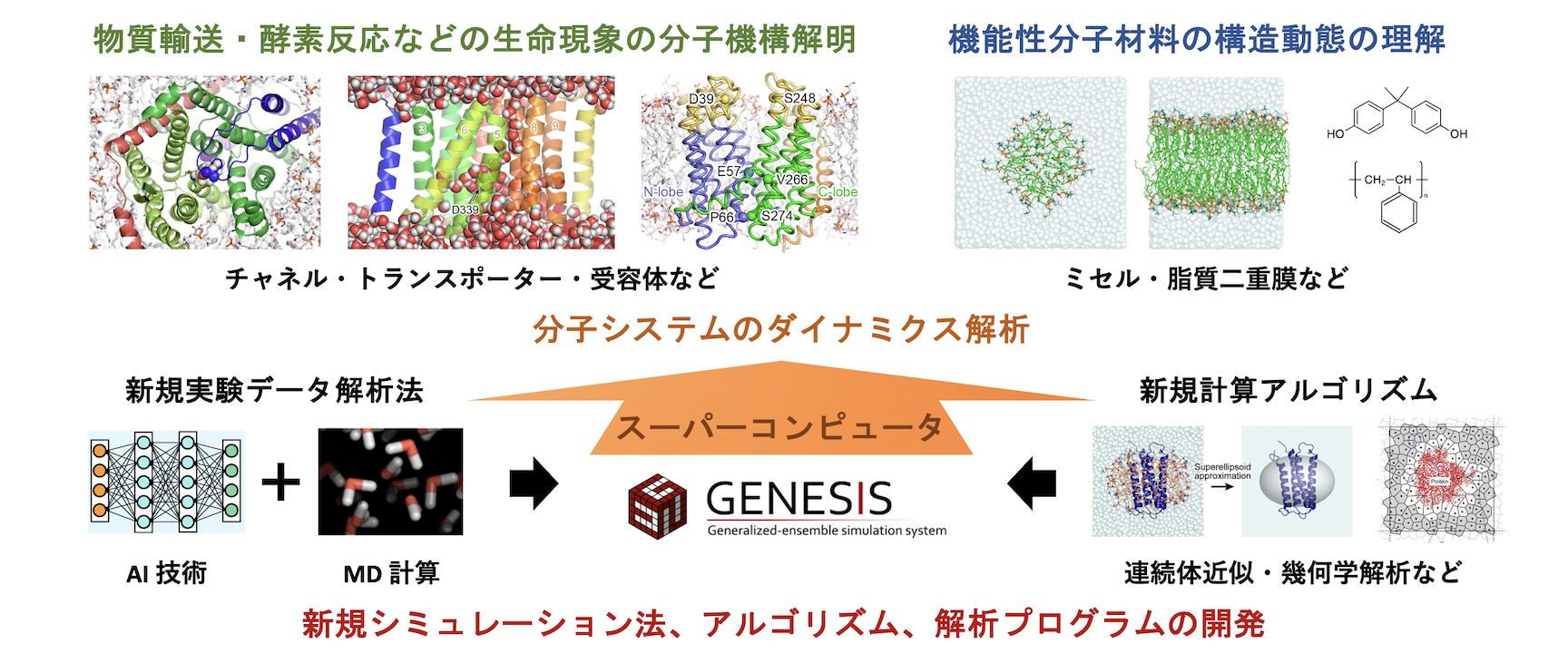
多くの分子はその構造や状態変化に起因する機能を有します。例えば、生体分子の1つであるタンパク質は、構造を変化させることによって、細胞内で物質輸送や酵素反応などの生命機能を発揮します。本研究室では、タンパク質・脂質・核酸・糖鎖などの生体分子を対象として、スーパーコンピュータを駆使しながら、主に「分子動力学シミュレーション」に基づいて分子の構造動態を理論予測し、可視化することで、分子機能の発現機構を原子レベルで理解することを目指しています。また、量子化学計算による機能性分子の構造解析、MD 計算と AI 技術を組み合わせた実験データ解析法の開発、抗がん剤や抗生物質とタンパク質との相互作用解析などにも取り組んでいます。
分子動力学シミュレーションとは?
『分子動力学シミュレーション』という言葉を、高校化学の教科書のコラムで見かけたことがある方もいるかもしれません。理論化学研究では、古典力学に基づく分子動力学計算(Molecular Dynamics; MD)や、量子力学に基づく量子化学計算(Quantum Mechanics; QM)が広く用いられます。QM 計算は分子の「電子状態」に焦点を当て、電子の挙動やエネルギー準位を解析する方法であるのに対して、MD 計算は「分子の動き」をシミュレーションし、分子間相互作用や構造変化などのダイナミクスを調べる方法です。MD シミュレーションでは、まず、分子システム中の各原子に働く力 F を、ファンデルワールス相互作用やクーロン相互作用などに基づいて数値的に計算します。力 F がわかれば、ニュートンの運動方程式 ma = F から加速度 a が求められ、その結果、原子の位置、速度、および加速度から原子が動く方向を予測できるようになります。このように、「ある瞬間における原子に働く力を計算し、原子の未来の位置を予測する」という計算を、コンピュータを使って非常に短い時間刻み(数フェムト秒)で数百万〜数億ステップ繰り返すことで、分子集団系のナノ秒からマイクロ秒オーダーの動きを追跡することができます。また、分子の構造や状態の変化を、統計熱力学に基づいてエネルギー論的に理解することもできます。MD シミュレーションは、原子間相互作用の精密な予測が必要な医薬品開発や材料開発などの産業界においても幅広く応用されています。

さて、突然ですが、「ラプラスの悪魔」をご存知でしょうか。これは1812年にフランスの数学者ピエール=シモン・ラプラスによって提唱された概念的存在で、ラプラスは「もしもある瞬間における全ての物質の力学的状態と力を知ることができ、かつそれらのデータを解析できるだけの知性が存在するならば、その知性にとっては、不確実なことは何もなく、未来も全て見通せるであろう」と提唱しました。MD シミュレーションは、近代の科学技術の進歩によって生み出された一種の「ラプラスの悪魔」とも言えるかもしれません。私たちは、MD シミュレーションを通じて、コンピュータ内で分子の世界が「未来」へ向かって刻一刻と変化する様子を観察できます。実際に MD 計算を行うと、分子は予想外の動きを見せることもあり、複雑な分子システムの挙動が単純ではないことに驚かされます。みなさんは、上図内のタンパク質分子やその周囲にある水分子が、この後どのように動いていくか想像できますでしょうか。興味を持った方は、ぜひ私たちと一緒に、MD シミュレーションを用いた研究に挑戦してみませんか?
研究室の主要テーマと具体的な研究例
生命機能の分子メカニズムの解明
細胞内で起こる生命現象は、タンパク質や脂質・核酸・糖鎖などの生体分子の働きによって精巧に制御されています。例えば、細胞膜を隔てたイオンや代謝物などの物質輸送は、チャネルやポンプ、トランスポーターと呼ばれる、細胞膜に「通り道」を作って物質の選択的透過を行う膜タンパク質の開閉運動によって実現されています。このような運動は、マイクロ秒〜ミリ秒オーダーで起こることが分かっていますが、実験では最先端の顕微鏡を以ってしても原子解像度で直接観察することが難しいため、計算化学に基づいたコンピュータ・シミュレーションによる解析が広く行われます。本研究室では、世界トップクラスの
スーパーコンピュータ「富岳」や、研究室所有の GPU 搭載クラスター計算機などを駆使しながら、MD シミュレーションを用いてタンパク質の構造動態を理論予測し、可視化することで、タンパク質の作動原理や機能発現メカニズムを理解することを目指しています。また、研究室独自の研究テーマに加えて、学内外の構造生物学や生化学の実験グループとも共同研究を進めています。
細菌や植物は、自然界に存在する無機硫黄化合物を細胞内に取り込み、それらをシステインやメチオニンなどの有機硫黄化合物に変換して生命機能の維持に利用しています。無機硫黄化合物を細胞内に取り込む過程において、細胞膜に存在する硫黄化合物トランスポーターが重要な役割を果たしています。私たちは、チオ硫酸イオントランスポーター YeeE/YeeD 複合体を対象として、スーパーコンピュータを用いた MD シミュレーションにより脂質二重膜中での分子動態を解析したところ、チオ硫酸イオン輸送過程において YeeD のシステイン (Cys17) の側鎖がチオ硫酸イオンによって修飾を受けると、輸送の完遂に必要不可欠な YeeD の揺らぎの増大が起きることを観察することに成功しました(奈良先端科学技術大学院大学・塚崎グループとの共同研究) (M. Ikei
et al.,
PLOS Biology (2024))。
その他の主要テーマについて
順次紹介する予定です。
参考資料
- "Elucidation of interactions regulating conformational stability and dynamics of SARS-CoV-2 S-protein",
T. Mori, J. Jung, C. Kobayashi, H. M. Dokainish, S. Re, and Y. Sugita,
Biophys. J., 120, 1060–1071 (2021).
- "GENESIS: A hybrid-parallel and multi-scale molecular dynamics simulator
with enhanced sampling algorithms for biomolecular and cellular simulations",
J. Jung, T. Mori, C. Kobayashi, Y. Matsunaga, T. Yoda, M. Feig, and Y. Sugita,
WIREs Comput. Mol. Sci., 5, 310–323 (2015).
- "膜タンパク質の全原子分子動力学シミュレーション",
森貴治, 松永康佑, 杉田有治,
膜タンパク質工学ハンドブック, 第3章 第2節, 津本浩平・浜窪隆雄 監修 (2020).
- "スーパーコンピュータを用いた膜タンパク質の分子動力学シミュレーション",
杉田有治, 森貴治, A. Pisliakov,
膜タンパク質構造研究, 第26章, 岩田想 編 (2013).
 東京理科大学 理学部第一部化学科 森研究室
東京理科大学 理学部第一部化学科 森研究室

